Chip sequenciador decodifica DNA próton por próton
Redação do Site Inovação Tecnológica - 22/07/2011

[Imagem: Ion Torrent]
Um chip capaz de sequenciar um genoma inteiro.
A nova tecnologia foi apresentada pela empresa Ion Torrent, em um artigo de seus pesquisadores, publicado na revista Nature.
Lei de Moore para a genética
Além de três bactérias, o chip foi testado para sequenciar o genoma de um humano. Especificamente, de um humano chamado Gordon Moore, autor da famosa Lei de Moore, que estabelece que o número de transistores dentro de um chip dobra a cada 18 meses.
Nenhuma outra jogada de marketing conseguiria atrair tamanha atenção, mesmo com a importância de uma tecnologia de sequenciamento genético baseada em um chip.
A Ion Torrent afirma que sua tecnologia permitirá que o sequenciamento do genoma humano custe US$1.000,00 nos próximos dois anos.
Tudo baseado na Lei de Moore: se a capacidade do chip sequenciador seguir o mesmo ritmo dos microprocessadores, a empresa calcula que bastará uma única geração de melhorias para que o sequenciamento genômico fique ao alcance de qualquer bolso.
Hoje, o chip sequenciador já custa apenas US$99,00. Mas o equipamento completo para fazer o sequenciamento, chip incluído, custa cerca de US$50.000,00.
[Imagem: Ion Torrent]
Tecnologias de sequenciamento genético
O fato é que a tecnologia baseada em semicondutor representa um avanço substancial em relação às atuais tecnologias de sequenciamento genético, ainda que não tenha atingido o mesmo nível de precisão das máquinas muito mais caras.
Os sequenciadores genéticos atuais usam detecção óptica. Uma fita única de DNA é convertida em uma fita dupla fazendo com que a segunda fita cresça base por base. Com o uso de marcadores, essas bases podem ser detectadas por tecnologia óptica, resultando na sequência genética.
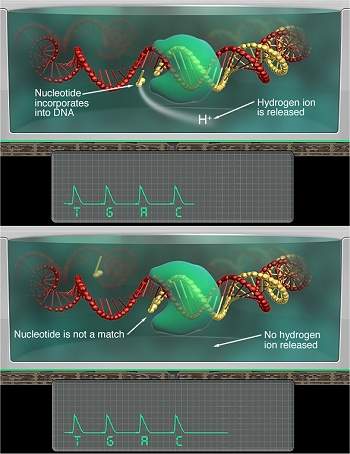
O chip sequenciador é muito mais simples. Em vez dos reagentes para marcar as bases, o chip detecta uma elevação no pH que ocorre conforme cada nucleotídeo se junta à fita em crescimento e libera um próton (H+) no processo.
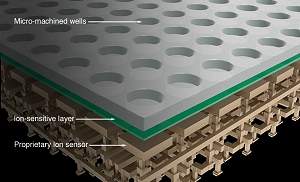
O chip contém uma malha de nanofuros, cada um contendo um modelo diferente de DNA, uma espécie de gabarito. Abaixo desses furos vem uma camada capaz de detectar os prótons e, abaixo, o sensor principal do chip. É por isto que a empresa afirma que seu chip lê o genoma próton por próton.
"Se um nucleotídeo, por exemplo um C, é adicionado a um dos gabaritos [nos nanofuros] e então incorporado à fita de DNA, será liberado um íon de hidrogênio. A carga do íon altera o pH da solução, o que pode ser detectado por nosso sensor de íons. Nosso sequenciador - essencialmente o menor peagâmetro de estado sólido do mundo - vai nomear a base, passando diretamente da informação química para a informação digital," explica a empresa.
Uma comparação bastante elucidativa pode ser feita com um sensor CCD de uma câmera digital. Enquanto o CCD capta fótons para registrar as imagens, o sensor do chip sequenciador de DNA "capta uma reação química". Como o nucleotídeo já está identificado, a informação do genoma é registrada diretamente em formato digital.
Há outras técnicas de sequenciamento genômico mais futurísticas, ainda em fase de desenvolvimento, como o sequenciamento eletrônico do DNA.
[Imagem: Ion Torrent]
Maior resolução
Outros pesquisadores, não envolvidos no desenvolvimento do chip sequenciador, afirmam que sua precisão ainda deixa a desejar.
A empresa parece saber bem disso, e demonstra esperar o concurso da Lei de Moore para resolver o problema: ocorre que, grosso modo, a densidade dos nanofuros, onde ficam as fitas individuais de DNA que servem de modelo, define a precisão da medição - quanto mais nanofuros, maior será a precisão.
Continuando com a analogia com o sensor de uma máquina digital, é como se o sequenciamento de DNA mais preciso dependesse agora de alguns "megapixels" a mais - na verdade, de um adensamento da malha de nanofuros e das respectivas fitas-modelo de DNA.
Agora é só esperar para ver se a Lei de Moore aplica-se também ao mundo da biologia.
Artigo: An integrated semiconductor device enabling non-optical genome sequencing
Autores: Jonathan M. Rothberg et al.
Revista: Nature
Data: 20 July 2011
Vol.: Published online
DOI: 10.1038/nature10242


Descoberta primeira molécula fractal da natureza
Impressora 3D se reconfigura para imprimir com tintas desconhecidas
A vida pode ser baseada em uma violação da lei física de ação e reação?
Você não precisa de cola para unir materiais - eletricidade basta
Lançada tecnologia de código aberto para interface com neurônios vivos
Objetos de madeira são feitos por impressão 3D
Inovação matemática melhora muito detecção de terremotos
Melhor rota para naves espaciais é traçada com teoria dos nós
Monociclo com duas pernas será ideal para fazer entregas
Bateria de ferro totalmente líquida armazena energias renováveis
Fotografado pela primeira vez bizarro cristal formado apenas por elétrons
Criados bits quânticos que mantém dados à temperatura ambiente
Expansão do Universo pode estar desacelerando
Detectada onda gravitacional que pode ajudar a resolver mistério cósmico
Resolvido antigo mistério sobre a formação dos cristais
Único no mundo, chip que funciona com luz é lançado industrialmente
Todos os direitos reservados.
É proibida a reprodução total ou parcial, por qualquer meio, sem prévia autorização por escrito.