Biochip permite sequenciar moléculas grandes de DNA
Redação do Site Inovação Tecnológica - 11/01/2010
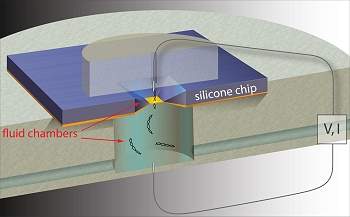
[Imagem: Nature Nanotechnology]
Pesquisadores da Universidade de Boston, nos Estados Unidos, desenvolveram uma tecnologia para sequenciar genomas mais rapidamente e com menor custo.
A novidade reduz dramaticamente a quantidade de DNA necessária para o sequenciamento, eliminando a fase de amplificação de DNA, que é cara, demorada e sujeita a erros.
No buraco da agulha
A técnica consiste em detectar moléculas de DNA à medida que elas passam por nanoporos de silício no interior de um biochip.
Campos elétricos são usados para inserir longas fitas de DNA através de poros cujos diâmetros medem apenas 4 nanômetros de largura, de forma parecida com passar uma linha pelo buraco de uma agulha.
A seguir, medições ultra sensíveis das correntes elétricas que atravessam o biochip são usadas para detectar cada molécula de DNA à medida que ela passa pelo nanoporo.
Fotocópias moleculares
"A fase atual da pesquisa indica que podemos detectar quantidades muito menores de amostras de DNA do que se havia conseguido antes. Quando pesquisadores começarem a implementar o sequenciamento de genomas ou a obtenção de perfis genômicos usando nanoporos, eles poderão usar nossa abordagem para reduzir enormemente o número de cópias usadas nessas medições", disse Amit Meller, coordenador do grupo.
Atualmente, o sequenciamento de genomas usa a amplificação de DNA para fazer bilhões de cópias moleculares, criando uma amostra grande o suficiente para ser analisada. Além do tempo e do custo desse processo, algumas das moléculas - que são como fotocópias de fotocópias - não saem perfeitas.
Fitas de DNA mais longas
A nova técnica usa campos elétricos circulando pelo bocal de entrada dos nanoporos para atrair fitas grandes de DNA, com carga elétrica negativa, fazendo-as passar pelos orifícios, onde a sequência de DNA pode ser lida. Como o DNA é atraído para os nanoporos a distância, poucas cópias da molécula são necessárias para que a análise possa ser feita.
"As tecnologias atuais de amplificação de DNA limitam o comprimento da molécula a ser utilizada a menos de mil pares de base. Como nosso método elimina a amplificação, ele não apenas reduz o custo, o tempo e a taxa de erros como também permite que a análise seja feita em fitas de DNA muito longas, muito maiores do que as usadas atualmente," afirmou Meller.
Espaguete e nanotecnologia
A técnica se tornou possível graças a uma descoberta surpreendente feita pelas cientistas: quanto mais longa a fita de DNA, mais rapidamente ele "encontra" a abertura do nanoporo.
"Isto é realmente surpreendente. Seria de se esperar que, se você tem um 'espaguete' mais longo, então achar sua ponta seria muito mais difícil," diz Meller. Contudo, como virtualmente tudo em nanoescala, a eletrofísica das dimensões nanométricas também apresenta regras e comportamentos que contrariam a experiência do mundo macroscópico.
Artigo: Electrostatic Focusing of Unlabelled DNA into Nanoscale Pores Using a Salt Gradient
Autores: Meni Wanunu, Will Morrison, Yitzhak Rabin, Alexander Y. Grosberg, Amit Meller
Revista: Nature Nanotechnology
Data: 20 December 2009
Vol.: Published online before print
DOI: 10.1038/nnano.2009.379


Corrosão inesperada: Os riscos de lixar o aço inoxidável
Exames de ultrassom agora em cores e muito mais detalhes
Blindagem eletromagnética pode ser instalada em janela transparente
Computação com luz é ótimo, mas como fazer a luz pensar em números negativos?
Bateria de ferrugem melhora com o tempo, guardando cada vez mais carga
Energia das ondas é extraída com eficiência ótima usando giroscópios
Um feixe de luz vira dois com características completamente diferentes
Processador fotônico roda por horas a temperatura ambiente
Armazenar dados em DNA é mais fácil do que se pensava
Tempo quântico pode ser medido sem um relógio
Barreiras de luz controlam fluxo de fluido em três dimensões
Iontrônica: Chip nanofluídico tem memória semelhante à do cérebro
Chip filma e controla reação química em tempo real
Esculpir a água permite formar relevos líquidos
Girinos ciborgues recebem implantes que se incorporam ao seu cérebro
Manto da invisibilidade magnético torna caminhos invisíveis
Todos os direitos reservados.
É proibida a reprodução total ou parcial, por qualquer meio, sem prévia autorização por escrito.